Transcription chez les eucaryotes
Le principe de la transcription est toujours le même : il s'agit de transcrire un code d'un système à quatre lettres (les bases nucléiques) dans un autre "langage" à 4 lettres, mais les modalités diffèrent par rapport aux procaryotes.
Localisation de la transcription
La synthèse des ARN a lieu là où se trouve l'ADN, c'est-à dire principalement dans le noyau des eucaryotes, mais aussi dans le nucléoïdes des mitochondries ou des chloroplastes.
De plus, l'ARN messager des eucaryotes qui est transcrit dans le noyau, subit ensuite une maturation, puis il est transféré dans le cytoplasme avant d'y être traduit.
Les ARN polymérases
Alors qu'une seule enzyme synthétise toutes les catégories d'ARN des procaryotes, dans la cellule eucaryotique, trois ARN polymérases sont mises en jeu dans la transcription d'ensembles de cistrons différents.
L'ARN polymérase A transcrit les cistrons ribosomiques, elle assure la synthèse des plus grands ARN des ribosomes. Son lieu d'action est le nucléole, en effet, les très nombreux cistrons ribosomiques répétés (c'est une de leur caractéristique), sont groupés au niveau de loci précis : les organisateurs nucléolaires. A l'interphase, la transcription active de ces cistrons se manifeste sous forme de structures cytologiques : les nucléoles.
L'ARN polymérase B réalise la synthèse de tous les ARN messagers nucléaires, qui seront traduits en protéines.
L'ARN polymérase C assure la synthèse des petits ARN nucléaires (ARN de transfert et ARN ribosomique 5S) et la transcription des gènes cytoplasmiques, c'est à dire, ceux qui sont portés par les chromosomes des mitochondries et des plastes des végétaux.
Toutes ces polymérases sont des édifices protéiques complexes composés de quatre sous unités, rappelant celles des procaryotes, auxquelles s'ajoutent jusqu'à une douzaine de protomères. De plus, ces complexes ne fonctionnent in vivo que grâce à l'interaction de nombreux autres facteurs protéiques établissant des relations temporaires avec le cœur de l'enzyme.
Ce que l'on connaît de la transcription des cistrons ribosomiques rappelle la transcription de l'ADN procaryotique et nous ne nous y attarderons pas.
La transcription des cistrons exprimés en protéines
La transcription des séquences codantes, les cistrons exprimés en protéines, est assurée par la polymérase B.
Parmi ceux-ci, on distingue plusieurs types d'organisation : quelques rares gènes présentent des unités de transcription semblables à celles des procaryotes, cependant la plupart des gènes eucaryotes sont structurés en mosaïque de portions codantes (les exons) et de portions n'ayant pas de signification protéique (les introns).
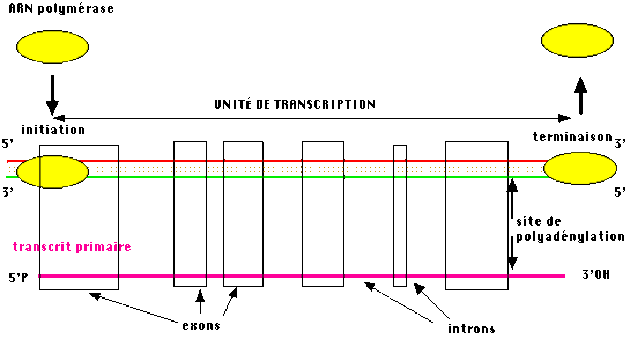
L'organisation générale d'un messager eucaryotique est présentée ci-dessous :
La synthèse d'ARN chez les eucaryotes donne généralement naissance à un produit de transcription "primaire" (ou prémessager) qui devra subir une maturation pour fournir le messager cytoplasmique fonctionnel.
Nous allons donc distinguer les deux étapes dans la "fabrication" d'un ARN messager fonctionnel : la transcription proprement dite et la maturation.
Le mécanisme de la transcription chez les eucaryotes
L'étape d'initiation de la transcription est cruciale, bien plus que l'élongation et à la terminaison, elle présente des caractéristiques propres aux eucaryotes, elle est décrite ci-dessous.
La transcription des séquences codantes est assurée par la polymérase B, mais d'autres facteurs protéiques (appartenant au groupe des TF II) sont nécessaires pour certaines étapes particulières, notamment pour le démarrage (l'initiation).
La formation du complexe d'initiation nécessaire à la fixation précise de l'ARN polymérase B est résumée dans la figure ci-contre.
Un premier complexe, TFII-D reconnait le promoteur et permet la fixation de TFII-A puis des interactions protéiques entre cet assemblage, TFII-B et la polymérase B permettent la fixation de celle-ci, son maintien est assuré par TFII-E.
D'autres facteurs (TFII-H et J) participent à la modification de la topologie de l'ADN .
C'est au prix de cette complexité que la transcription est correctement initiée !
Ici encore, tout repose sur l'interaction de protéines spécifiques avec des séquences déterminées, les promoteurs.
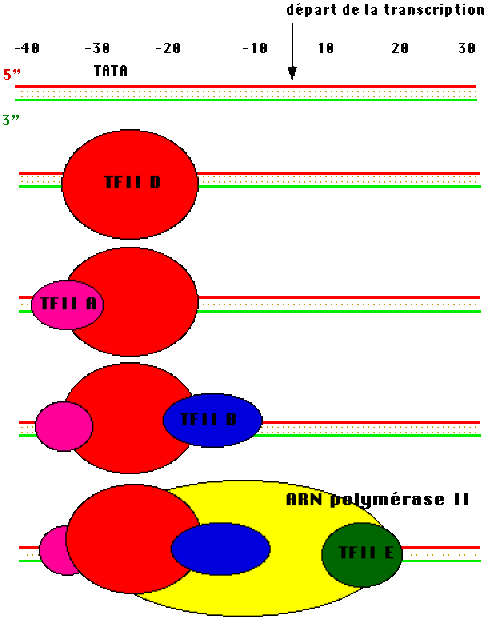
Les promoteurs des eucaryotes, reconnus par l'ARN polymérase B (ou II), ont été étudiés à l'aide des outils les plus performants de la génétique moléculaire. Des expériences ont permis l'analyse systématique de l'effet de mutations dans les régions situées en amont du site de démarrage de la transcription. Leurs résultats ont mis en évidence le rôle déterminant de trois 'modules', positionnés à des distances d'environ -30, -75 et -100 du point de démarrage de la transcription :
La cassette TATA, vers -30, est probablement la seule séquence consensus située à une position fixe présente dans la quasi totalité des gènes de la classe II, elle semble jouer un rôle dans la précision du premier nucléotide transcrit.
La cassette CAAT est située à -80 dans l'exemple du gène de la globine mais sa position peut varier selon les gènes, séquence consensus : GGCCAATCT.
La cassette GC (consensus : GGGCGG) est souvent en plusieurs copies et dans les deux orientations.
Ces modules de base représentent donc un signal pour le complexe d'initiation de la polymérase B mais leur assemblage en différentes combinaisons possibles permet déjà une diversification qui va intervenir dans la régulation de la transcription.
D'autres modules, parfois très éloignés du promoteur "de base", jouent également un rôle dans la régulation.
Les signaux de terminaison sont moins bien connus que chez les procaryotes, par contre on connait des signaux de polyadénylation (l'ajout d'une séquence polyadénylée en 3' OH des messagers eucaryotiques fait partie de la maturation).
La maturation
La maturation de la plupart des transcrits primaires porte sur 3 points :
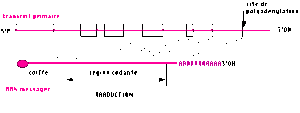
la formation d'une structure particulière en 5' : la coiffe,
l'adjonction d'une séquence polyadénylée en 3',
l'épissage : excision des introns et jonction des exons.
La coiffe est formée par addition d'une guanosine triphosphate : le premier nucléotide du messager est généralement une purine, A ou G et représente théoriquement l'extrémité 5' triphosphorylée de la molécule : 5'pppA-pN-pN-pN-pN-pN ...
En fait, une guanine est ajoutée par une liaison inhabituelle 5'-5' : Gppp + pppA-pN-pN-pN-pN-pN \(\rightarrow\) Gp-ppA-pN-pN-pN-pN-pN ...
Diverses méthylations peuvent se produire ensuite, notamment une en position 7 de la guanine et sur le ribose pour compléter cette structure que l'on retrouve dans tous les messagers eucaryotiques.
L'adjonction de la coiffe se fait au cours de la synthèse du transcrit primaire. La coiffe joue un rôle pour le démarrage de la traduction : elle permet la reconnaissance, par les ribosomes, de l'extrémité 5' de l'ARN messager.
La polyadénylation est un ajout post-transcriptionnel de nucléotides adényliques au niveau d'un site de polyadénylation du transcrit primaire. Le site est reconnu par un complexe protéique comportant une poly-A polymérase.
Cette structure va former l'extrémité 3' du messager, elle peut aller jusqu'à 200 nucléotides. Elle semble assurer la protection de l'ARNm contre les dégradations enzymatiques et jouerait un rôle dans l'adressage du messager vers le cytoplasme.
La maturation des gènes mosaïques, qui sont transcrits depuis le point d'initiation jusqu'au signal de terminaison, assure l'élimination des introns du transcrit primaire, ou pré-messager, et la jonction des exons qui doit se faire avec une grande précision. L'ensemble se fait simultanément par un mécanisme d' "épissage".
Il existe plusieurs mécanismes d'épissage, qui diffèrent selon les unités de transcription. Tous font intervenir des molécules d'ARN autres que le transcrit primaire. Dans certains cas, c'est même un ARN qui catalyse la réaction : un ribozyme.
Le mécanisme décrit ici est celui utilisé pour la plupart des messagers nucléaires, il fait appel à des ribonucléoprotéines au sein desquels, la molécule d'ARN joue certainementun rôle prépondérant.
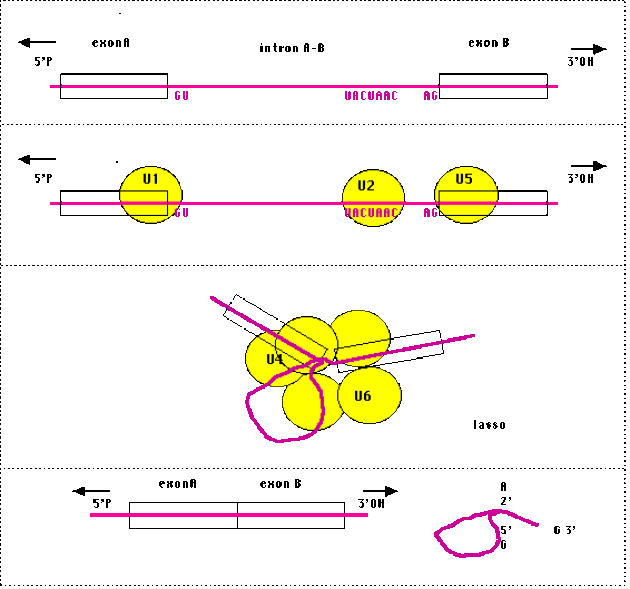
L'analyse de nombreux introns de levure a fait apparaitre trois séquences consensus que l'on peut généraliser à d'autres eucaryotes :
A l'extrémité 5' de l'intron : le consensus gauche est représenté par le dinucléotide GU
A la jonction entre l'extrémité 3' de l'intron et l'extrémité 5' de l'exon suivant : le dinucléotide AG, consensus à droite
Peu avant l'extrémité 3' de l'intron , la séquence de branchement, UACUAAC chez les messagers de levure et, plus généralement, Py N Py Py Pu A Py* chez les eucaryotes supérieurs est située et appelée pour les raisons évoquées ci-dessous.
* où Py représente une pyrimidine, N un nucléotide quelconque, Pu une purine, l'adénine étant remarquablement constante.
Des mutations dans les nucléotides codant pour ces trois séquences d'un intron empêchent l'épissage correct des exons qui l'encadrent. Ces séquences sont reconnues par des ribonucléoprotéines (en jaune sur le schéma) qui vont former un complexe nécessaire à l'épissage, visible en microscopie électronique, appelé spliceosome (de l'anglais splicing = épissage), snRNP ou snurp (Small Nuclear Uridine-rich RibonucleoProtein).
L'édifice représenté ci-contre, imposé par la liaison des différents snurps, provoque un repliement de l'intron qui permet une liaison phosphodiester entre l'extrémité 5' de l'intron et l'hydroxyle 2' de l'adénine de la séquence consensus "de branchement".
Une coupure intervient ensuite entre les extrémités 3' de l'intron et 5' de l'exon suivant et les deux exons sont reliés par une liaison phosphodiester.
Remarque :
*Le branchement en 2' de l'adénosine fait prendre une structure dite en "lasso" à l'intron. In vivo, ce lasso est immédiatement dégradé mais dans des expériences in vitro, on peut l'observer en microscopie électronique. Ce mécanisme complexe est génétiquement contrôlé et joue un rôle important dans la régulation de l'expression des gènes chez les eucaryotes.